C’est quoi la modélisation moléculaire?

Modélisation moléculaire et protéine
Connaître la structure 3D d’une protéine permet de comprendre son fonctionnement à l’échelle atomique, ses mouvements et comment elle interagit avec d’autres protéines ou d’autres molécules comme par exemple un médicament.
Les protéines sont trop petites (100 à 1’000 angströms) pour pouvoir être « vues » par des techniques de microscopie classiques.
La modélisation moléculaire permet de « visualiser » la structure 3D d’une protéine en s’appuyant entre autre sur la connaissance de la position de ses atomes.

Comment "voir" les atomes d'une protéine?
Voici 2 techniques qui permettent de déterminer «expérimentalement» la position des atomes d’une protéine.
La protéine est cristallisée puis le cristal est analysé grâce à la diffraction des rayons X.
La position de chaque atome peut ensuite être déterminée avec l’aide d’une analyse mathématique.
Cette technique existe depuis le début des années 1960.
Une analyse par Résonance Magnétique Nucléaire (RMN) de la protéine en solution, permet de déterminer la position relative des atomes d’hydrogène et de carbone, et indirectement la structure 3D de la protéine.
La cryo-microscopie électronique (cryo-ME) est une technique plus récente, qui permet d’étudier la structure 3D des protéines dans des échantillons biologiques congelés à -185°C.
Dans les années 1980, le Pr Jacques Dubochet a développé la vitrification, une méthode permettant de faire chuter la température de l’eau sans en changer la structure. Une découverte récompensée par le Prix Nobel de chimie, en 2017, qui est à la base de la cryo-ME. La structure 3D d’une hémoglobine (3.4 angstrom) a pu être déterminée à l’aide de cette technique (source).

La modélisation moléculaire: ça sert à quoi?
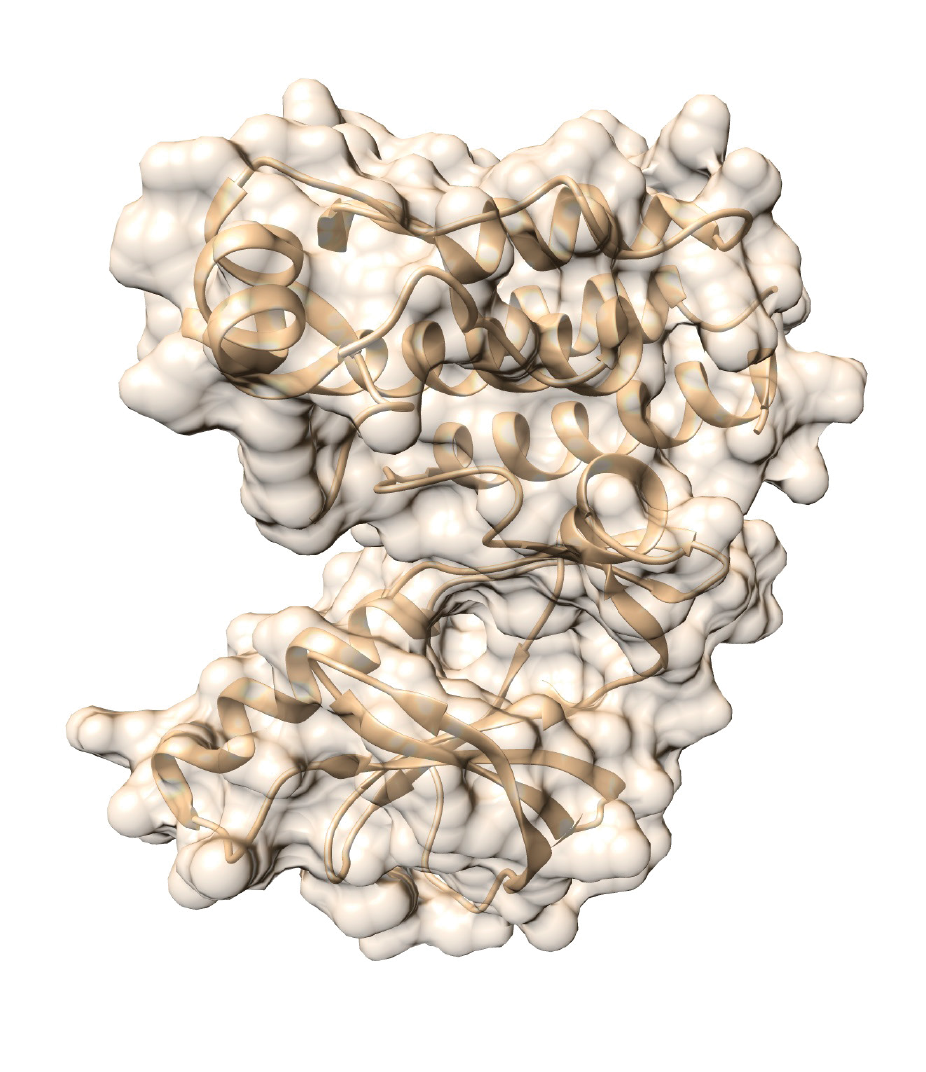
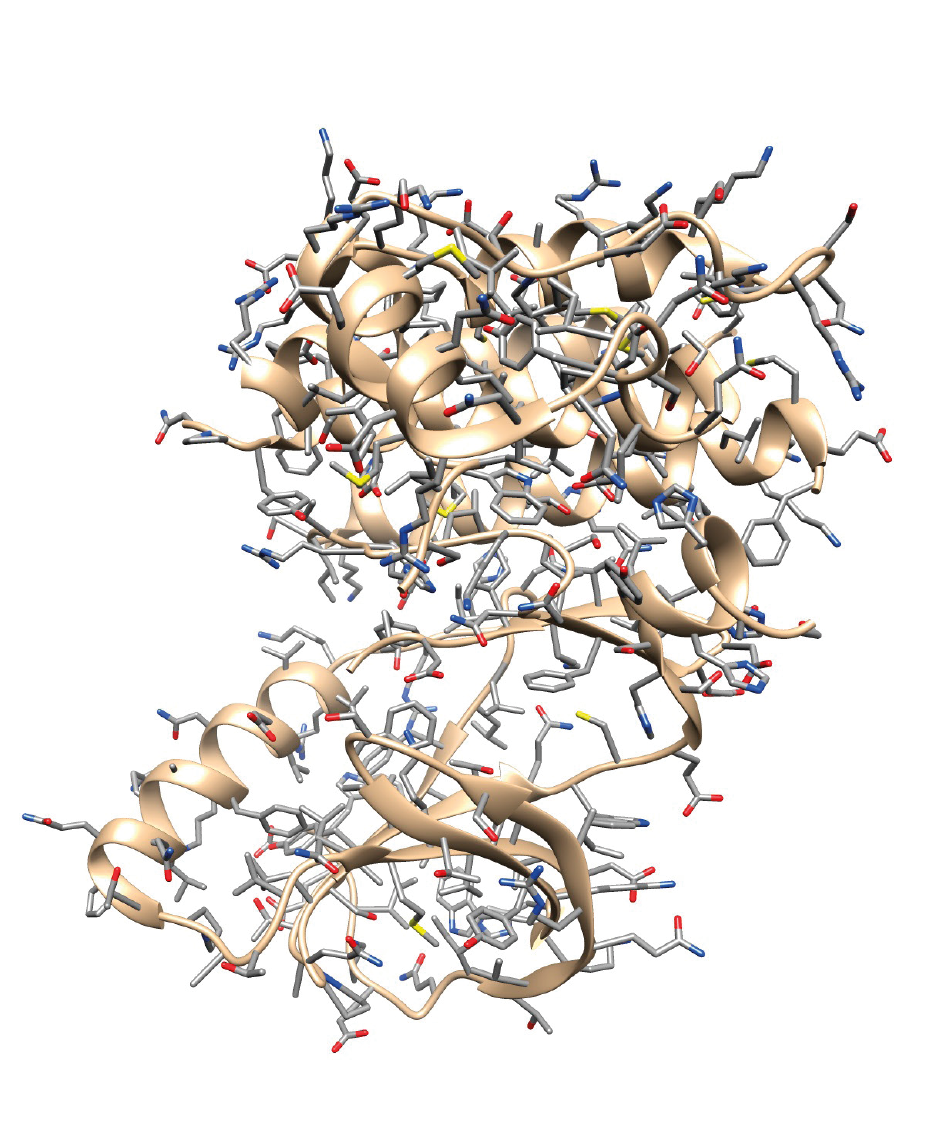
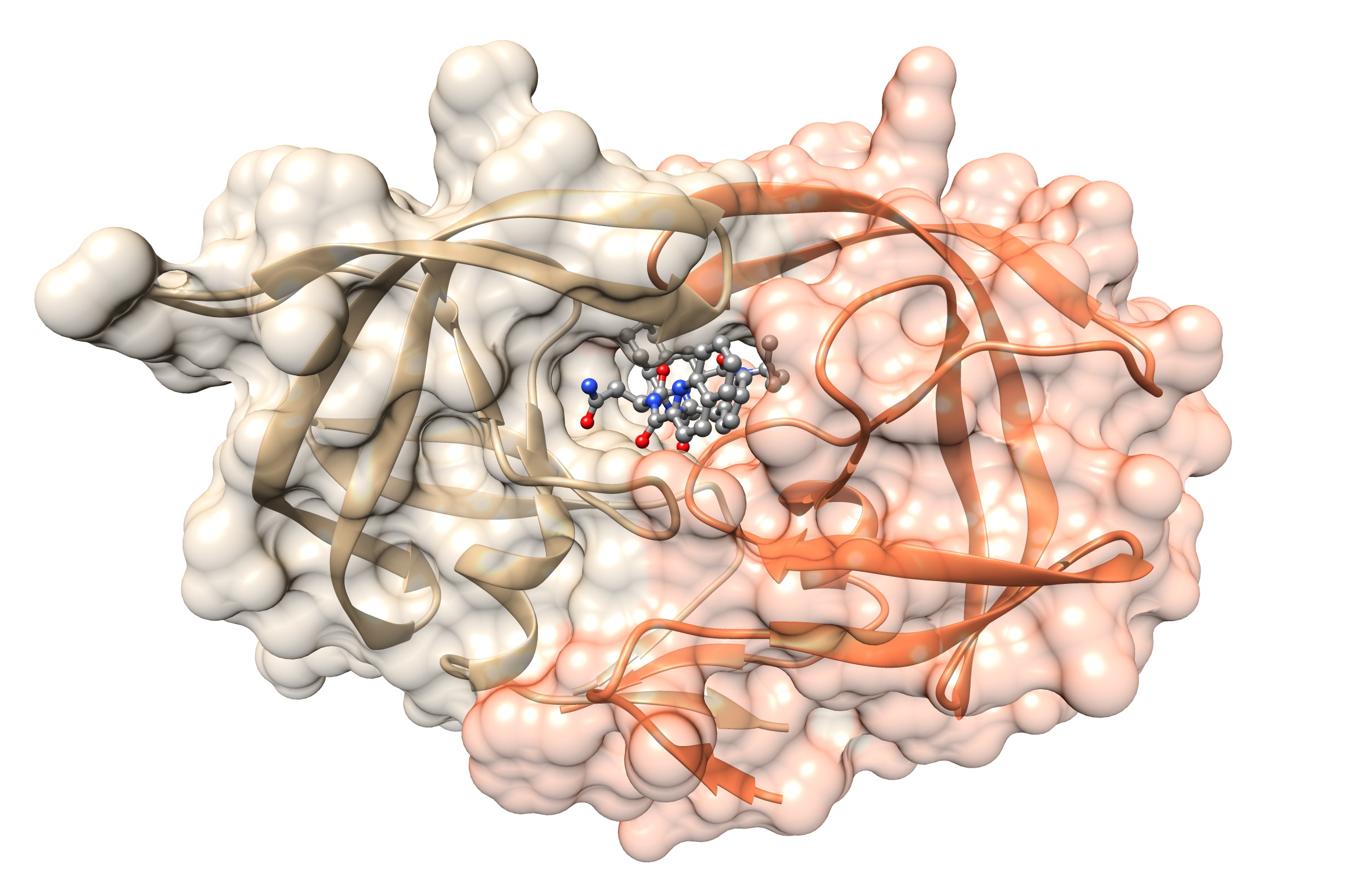
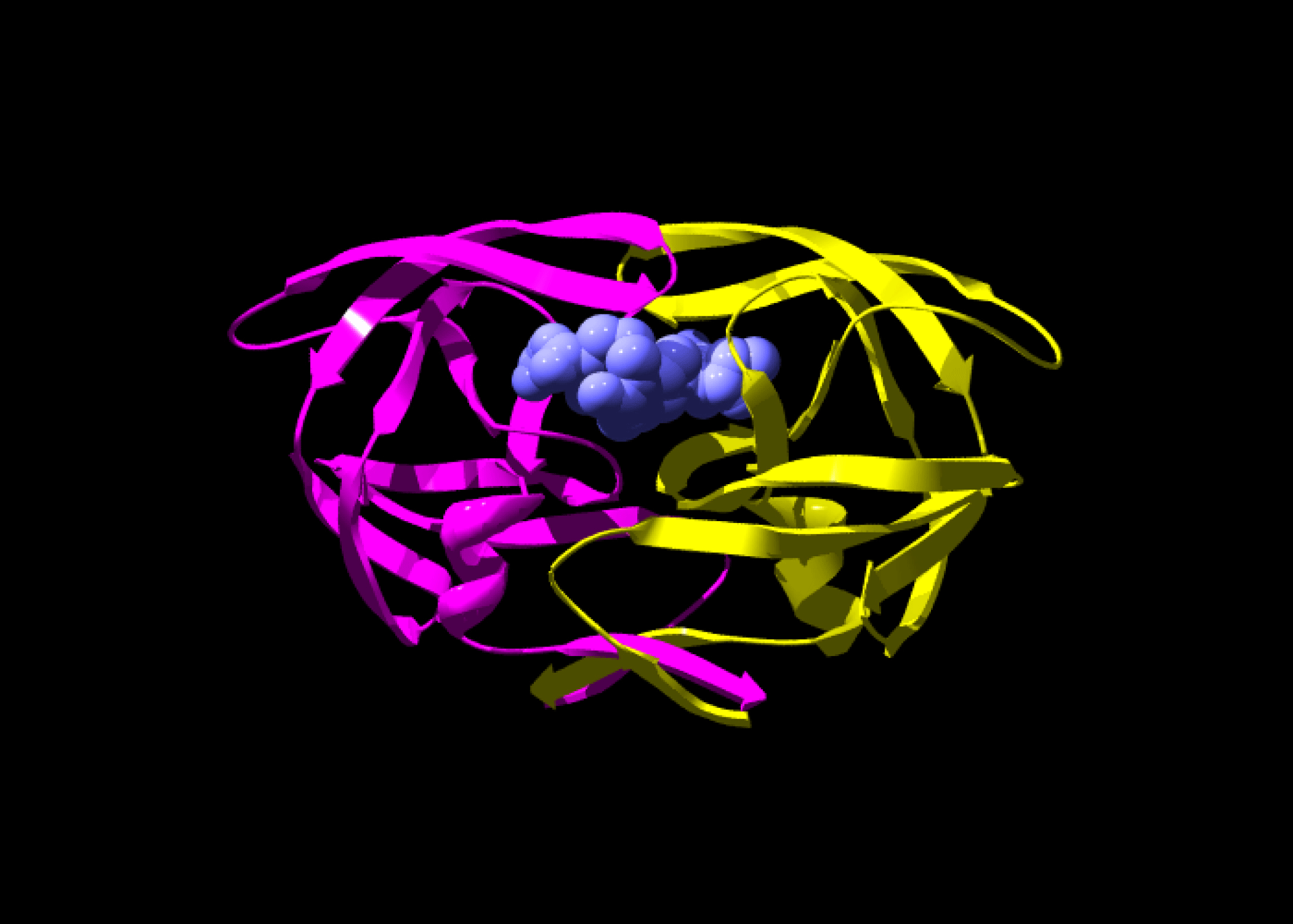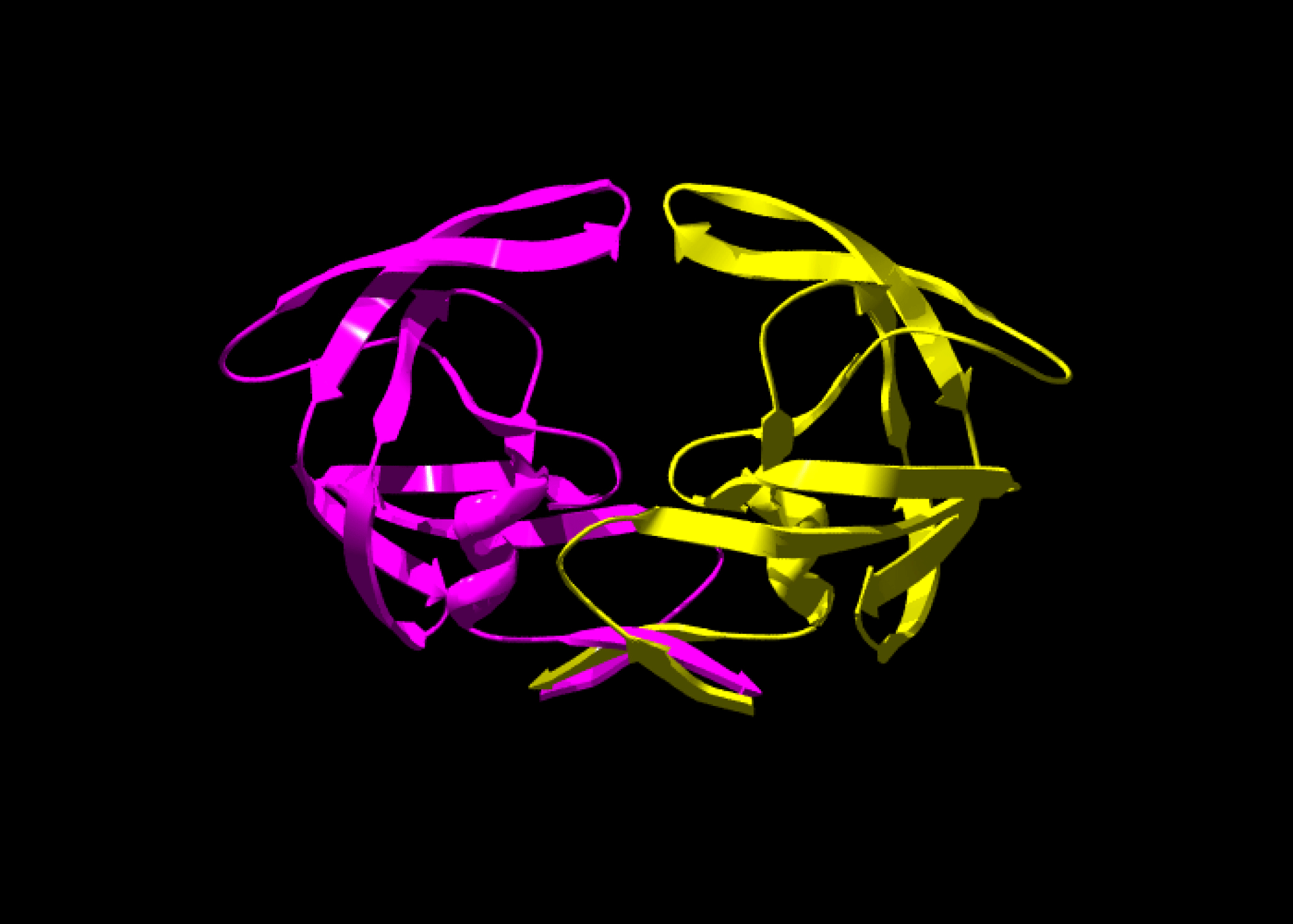
La modélisation moléculaire s’appuie sur la connaissance de la position des atomes et des liaisons entre les atomes de chaque acide aminé pour « dessiner » la structure 3D d’une protéine.
Voici 2 représentations de la structure 3D de la protéine BRAF qui est composée de 766 acides aminés (11’815 atomes).

La structure 3D d’environ 2/3 des protéines humaines n’a pas encore pu être déterminée «expérimentalement» (source UniProtKB).
La modélisation moléculaire permet de prédire la structure 3D de ces protéines en se basant sur la structure 3D déjà connue de protéines similaires.

La modélisation moléculaire permet de prédire l’effet d’un changement de un – ou plusieurs- acide(s) aminé(s) sur la structure 3D d’une protéine.
Il est ainsi possible de prédire l’effet d’une mutation sur l’activité d’une protéine et/ou sur sa capacité à interagir avec un médicament.
La modélisation moléculaire est utilisée, en particulier, lorsqu’aucune information biologique ou médicale n’est disponible sur une mutation.
C’est le cas pour environ 25 % des patients pris en charge par le Tumor Board Moléculaire du Réseau d’Oncologie Romand.
L'exemple BRAF V600E
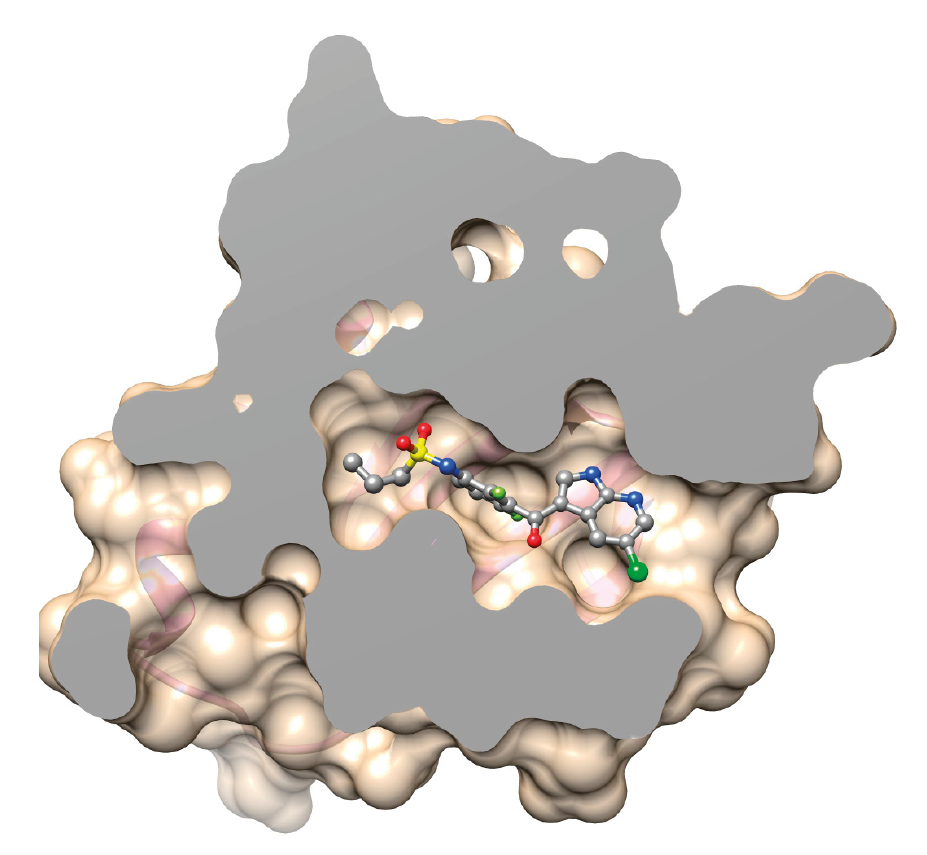
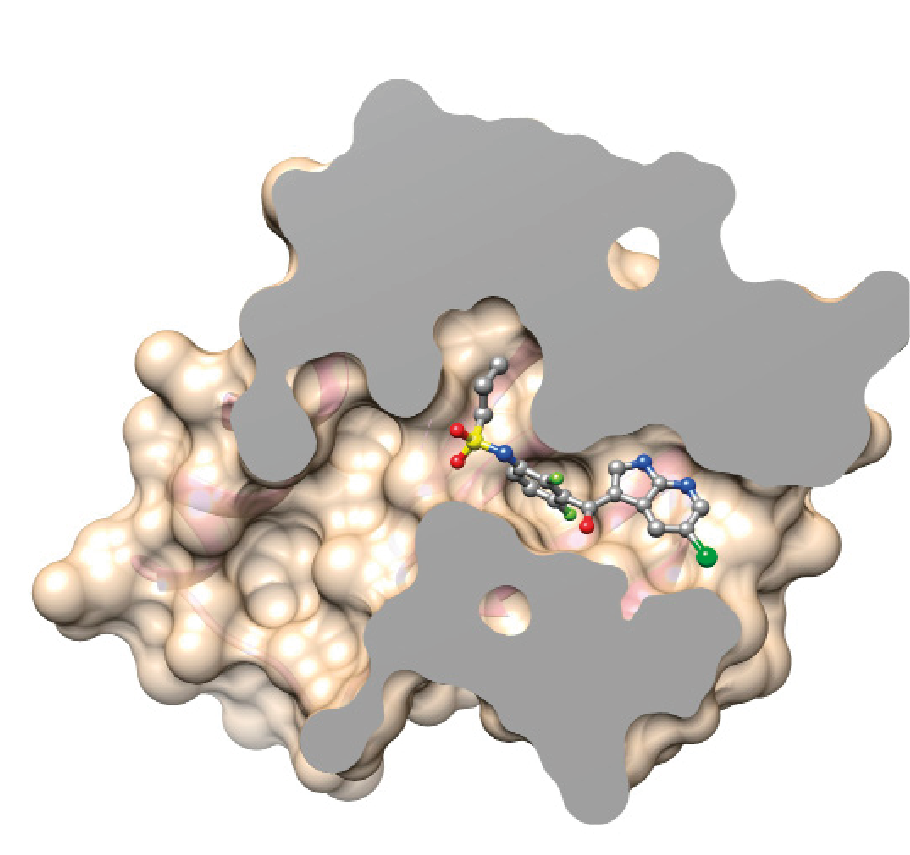
La protéine BRAF avec la mutation V600E n’est plus capable de contrôler la division des cellules. Le médicament vemurafenib cible la protéine mutée et bloque son activité.
La modélisation moléculaire permet de visualiser et de simuler l’effet de la mutation V600E sur l’interaction entre la protéine et le médicament.
Le médicament adopte une position qui est plus stable du point de vue biochimique et biophysique dans la protéine BRAF V600E. Le médicament a ainsi plus d’affinité pour la protéine BRAF V600E.
Le vemurafenib est utilisé pour traiter les patients dont les cellules cancéreuses ont la protéine BRAF avec la mutation V600E.
PLUS LOIN

« DRUG DESIGN »
Les techniques de modélisation moléculaire sont également utilisées pour concevoir de nouveaux médicaments.
Une fois que la structure 3D de la protéine que l’on souhaite cibler est connue, il est possible de «dessiner» un médicament susceptible de se loger dans le site fonctionnel de cette protéine. Le but est, le plus souvent, de bloquer l’activité de la protéine responsable de la maladie.
Un exemple historique: un médicament contre le SIDA
Mis sur le marché en 1987, le saquinavir est l’un des premiers médicaments conçu avec l’aide de programmes bioinformatiques.
Le saquinavir cible spécifiquement une des protéines du virus du SIDA, appelée protéase. L’activité de cette protéine est indispensable pour la réplication et la survie du virus.
Les chercheurs ont déterminé la structure 3D de la protéase. Ils ont ensuite «dessiné» un médicament susceptible de se loger dans le site fonctionnel de la protéase afin de bloquer son activité.
Et ça a marché !